Exploración y manipulación datos 👋
Nos empezamos a mover por los datos y a modificarlos
Navegación
Acceso por índice
Recordemos los objetos que manipularemos más frecuentemente.
En primer lugar trabajaremos con data frames df[ ], que por hacer un simil intuitivo son las bases de datos a las que estamos acostumbraados con otros programas como Excel. Los df están formados de filas y columnas, es decir, son objetos bidimensionales. Podemos acceder a los datos por su posición. Recordad que la sintaxis de un df se acompañaba de corchetes [ ] cuando queríamos acceder a sus elementos. Si indicamos solo un número dentro de los corchetes accederemos a la posición de la columna correspondiente, sin embargo, si colocamos una coma en medio del corchete, los índices que emplacemos a la izquierda se referiran a filas y aquellos de la derecha a columnas
mi_df #todo el df
mi_df[3] #tercera columna del df
mi_df[filas,columnas] # A la izquierda filas, a la derecha columnas
mi_df[1,] #primera observación con todas las columnas
mi_df[,1] #primera columna con todas las observaciones
Por otro lado existían los vectores. Los vectores eran cadenas de datos con solo una dimensión. Para crear vectores utilizabamos la función c() de concatenar. A pesar que son cadenas o tiras de datos de una dimensión, estos están ordenados y por tanato también podemos acceder a sus elementos por posiciones. Para acceder a la X posición de un vector es identíco que en los df, debemos referirnos al vector por su nombre acompañado de [ ] y el índice al que queremos acceder.
vector <- c(5,1,20)
vector [2] # Accedo al segundo elemento del vector, en este caso devolvería el número 1.
Ahora bien. tanto en los df como en los vectores hasta ahora solo hemos visto como acceder a un elemento en concreto, sin embargo, no os resultaría poco práctico si solo pudiesemos acceder individualmente a elementos?.
Creo un df de prueba, simulando la base de datos de una pequeña clase. Imaginaos que quiero acceder no solo a la variable de los nombres de los alumnos, sino que también quiero acceder al mismo tiempo a sus notas en el último parcial. En este caso estaría interesado en seleccionar a la vez dos columnas de mi df. Bien, pues para acceder a varios elementos simultaneamente puedo recurrir a vectores.
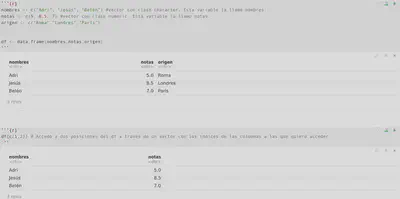
nombres <- c("Adri", "Jesús", "Belén") #vector con clase character. Esta variable la llamo nombres.
notas <- c(5, 8.5, 7) #vector con clase numeric. Esta variable la llamo notas.
origen <- c("Roma","Londres","Paris")
df <- data.frame(nombres,notas,origen)
df[c(1,2)] # Accedo a dos posiciones del df a través de un vector con los índices de las columnas a las que quiero acceder
Del mismo modo, puedo utilizar vectores para acceder a filas específicas de aquellas columnas que queramos.
df[c(1,2),c(3,1)] # Acceder a las filas 1 y 2 de las columnas 3 y 1. Fijad como puedo especificar también el orden
#resultado
# origen nombres
#1 Roma Adri
#2 Londres Jesús
# Podemos acceder con vectores tambien a las posiciones que queremos de otro vector
vector <- c(10,92,2,-4,91,-42,45,86,12,1)
vector[c(1,3,5)]
#resultado:
# [1] 10 2 91
Otro modo de crear accesos rápidos es a través de secuencias. En R hay un modo muy rápido para decir “todos los números desde el X hasta el Y”. Si quisiesemos acceder a los cinco primeros elementos de un vector podríamos escribir 1:5.
1:5 quiere decir un vector del 1 hasta el 5, del mismo modo, 5:1 sería el vector con dirección contraria, del 5 hasta el 1
vector <- c(10,92,2,-4,91,-42,45,86,12,1)
vector[1:5]
# resultado:
#[1] 10 92 2 -4 91
Aceso por nombre
Existe otro modo de acceder a columnas de un df. En vez de acceder a las posiciones de los objetos por sus índices, en los casos de los df podemos seleccionar las columnas por sus nombres. Todas las columnas tienen nombres, recordad que son las variables, y podemos llamarlas por su nombre.
La lógica es la misma que en la indexación, incluso podemos pasar vectores con los nombres de todas las variables que queremos seleccionar. Tal vez este modo pueda resultar más complejo que la indexación por número, sin embargo, imaginad un df con muchas variables … ¿es realmente práctico llamar por el orden de sus variables? En muchas ocasiones es más útil llamar a las variables por su nombre.
A veces es útil llamar a otra función que imprima los nombres de las variables de los df para saber como referenciarlas correctamente. Se puede utilizar la función names()
df["nombres"]
df[c("nombres","notas")] # se pueden crear vectores con nombres también
df[1,c("nombres,","notas")] # Incluso se puede segmentar por filas, igual que la indexación normal.
####
names(df) # La función names te mostrará los nombres de las variables de un df.
# Resultado: [1] "nombres" "notas" "origen"
Acceso a variables por $
Si queremos llamar a una variable dentro de un df directamente, podemos acceder mediante el comando $.
Las variables están dentro del df, por lo que de alguna manera las llamamos directamente si indicamos su “ruta”. En vez de escribir df[“nombres] podriamos escribir df$nombres .
Con este modo accedemos individualmente a las variabels, como si fuesen vectores. Es útil cuando queremos hacer transformaciones u operar solo con una variable en concreto.
Filtrar observaciones
Hay a veces que nos interesa filtrar las observaciones que cumplen una condición específica. Por ejemplo, si quisiese ver la lista de aprobados de una clase podría filtrar solo aquellas observaciones que en la columna “notas” tuviesen un cinco o más.
Lo que filtro son las observaciones por lo que la condición debe ir en la parte de las filas dentro de los corchetes del df.
# Voy a añadir una observación con suspenso al df.
df <- rbind(df,c("Antonio",4.5,"Boston"))
# nombres notas origen
#1 Adri 5 Roma
#2 Jesús 8.5 Londres
#3 Belén 7 París
#4 Antonio 4.5 Boston
df[df$notas >= 5,] # filtro las observaciones donde se cumpla la condición df$notas >= 5.
# nombres notas origen
#1 Adri 5 Roma
#2 Jesús 8.5 Londres
#3 Belén 7 París
########
vector
# [1] 10 92 2 -4 91 -42 45 86 12 1
vector[vector>5] # también puedo filtrar dentro de vectores
# [1] 10 92 91 45 86 12
TIDYVERSE
Existe una colección de paquetes muy extendida en R llamda tidyverse. Esta colección recoge paquetes que simplifican muchas tareas en R. Una de sus principales contribuciones viene dada por el paquete dplyr .
El paquete dplyr propone una nueva lógica y sintaxis para operar en R, incluyendo el modo en el que nos movemos por los objetos.
Introduzco el famoso pipe o %>% . El pipe es un modo de conectar operaciones con sintaxis dplyr. Lo necesitaremos para poder explotar al máximo las facilidades que puede proponer tidyverse y dplyr. Por simplificar su explicación el comando %>% conecta en orden varias instrucciones. La principal ventaja es que nos ahorra escribir muchos argumentos en las funciones. Veamos un ejemplo.
mean(df$notas) # Calculo la media del vector notas incluido en el df llamado df.
df$notas %>% # Lo mismo con lógica dplyr.
mean()
Tal vez con un ejemplo tan simple no se pueda obsevar todo el potencial que conlleva el comando %>% pero voy a hacer un pequeño salto en el tiempo para que podaís comparar a simple vista las ventajas de la lógica.
#Calcular la media de solo aquellos estudiantes que sean de Roma
mean(df[df$origen == "Roma","notas"])
...
Como había dicho, dplyr propone unas nuevas funciones para acceder a los objetos. Las funciones se llaman select() y filter().
Es una cuestión de gustos. No es obligatorio manejarse con la lógica de dplyr, sin embargo, en mi opinión es mucho más intuitiva que la sintáxis básica de R, sobre todo cuando más se avanza en complejidad.
select()
Con la función select() podemos seleccionar columnas de un modo sencillo las columnas
df[c("nombres","notas")]
df %>%
select(nombres,notas) #con dplyr y %>%
# o
select(df,nombres,notas) #con dplyr y sin %>%
filter()
Con la función filter() podemos filtrar observaciones de un modo sencillo
df[c("nombres","notas")]
df %>%
filter(origen == "Roma") #con dplyr y %>%
# o
select(df,origen == "Roma") #con dplyr y sin %>%
slice()
Con la función slice() podemos indicar con índices las filas que queremos conservar, parecido a cuando accediamos por índice a las filas con la sintaxis básica.
df %>%
slice(1,3)
# nombres notas origen
#1 Adri 5 Roma
#2 Belén 7 París
df %>%
slice_tail() # variación para ver la última fila
df %>%
slice_head() # variadción para ver la primera fula
Ejemplo práctico
Vamos a repasar los conceptos con un pequeño ejercicio práctico. Recuperemos la base de datos de pokemon del post anterior. Lo podeís descargar también aquí .
Partimos con la base de datos ya importada. Para este ejercicio he llamado al df pokemon.
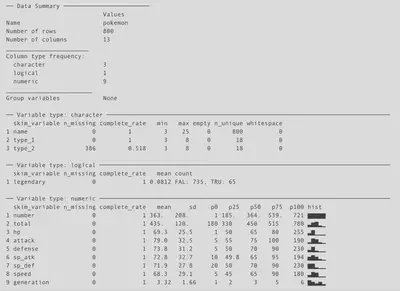
En primer lugar, y para acostumbrarnos, podemos ver la estructura y un resumen del df haciendo uso de la función skim()
Pongamos un ejercicio hipótetico (sobre la hipótesis previa de que los Pokémon existiesen).
Queremos echar un vistazo a los Pokémon de tipo fuego. Nuestro favorito es Charmander, de toda la vida, pero ahora estamos interesados en otros Pokémon de fuego parecido.
Como primera aproximación podríamos filtrar los Pokémon que fuesen de tipo fuego. Incluso podríamos crear un df que solo fuesen los tipo fuego.
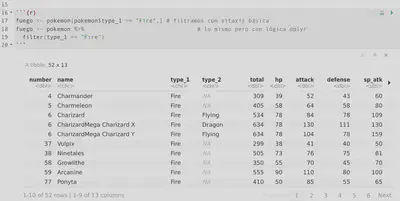
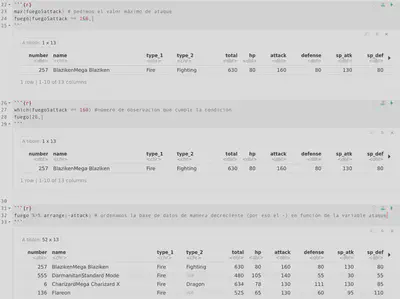
fuego <- pokemon[pokemon$type_1 == "Fire",] # filtramos con sitaxis básica
fuego <- pokemon %>% # lo mismo pero con lógica dplyr
filter(type_1 == "Fire")
Ahora que hemos filtrado solo los pokemon de tipo fuego podríamos seguir varias estrategias para ver quien tiene el mayor ataque. Dejo algunos modos, aunque hasta este punto lo principal es saber filtrar.
max(fuego$attack) # pedimos el valor máximo de ataque
fuego[fuego$attack == 160,]
###
which(fuego$attack == 160) #número de observacion que cumple la condición
fuego[26,]
###
fuego %>% arrange(-attack) # ordenamos la base de datos de manera decreciente (por eso el -) en función de la variable ataque
Filtrar con varias condiciones
Otro enfoque por el que podríamos optar es crear varias condiciones y anidarlas con los operadores lógicos de &(AND) u | (OR).
Por ejemplo: Filtrar solo los Pokémon de tipo fuego y que tengan más de 120 en ataque.
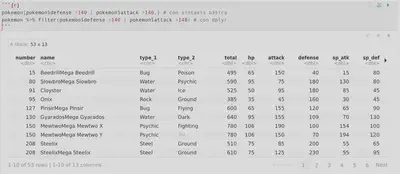
pokemon[pokemon$type_1 == "Fire" & pokemon$attack >120,] # con sintaxis básica
pokemon %>% filter(type_1 == "Fire" & attack > 120) # con dplyr
Por ejemplo: Filtrar los Pokémon que tengan más de 140 de defensa O que tengan más de 140 de ataque.
pokemon[pokemon$defense >140 | pokemon$attack >140,] # con sintaxis básica
pokemon %>% filter(pokemon$defense >140 | pokemon$attack >140) # con dplyr