Importación de datos y primera exploración de datos 👋
Trabajamos con nuestra primera base de datos
Una vez que hemos repasado los conceptos más básicos de R y su estructura básica es hora de trabajar con los primeros datos.
Sesión de Trabajo
Lo primero de todo, antes de pasar a importar cualquier dato, es tener en cuenta que R trabaja con sesiones y rutas de trabajo.
Por simplificarlo de alguna manera, una sesión de trabajo es una ventana de R. Un simil que se me puede ocurrir también es un navegador de internet. Cada ventana es una sesión abierta, que guarda historial, objetos, rutas etc… y puede contener más de una pestaña (scripts). Por lo general, en un uso básico de R solo tendremos una sesión abierta, pero teneís que saber que se pueden tener más de una sesión (o ventanas). Cada sesión es independiente y esto afecta tanto a los objetos que tengamos guardados como incluso a las librerias que hayamos activado. Si yo guardo un objeto, supongamos en la sesión A, no podré llamar a ese objeto en la sesión B ya que son totalmente independientes.
Ruta de Trabajo (Working Directory)
Tal vez lo más importante sobre las sesiones es que siempre necesita tener una “ubicación” en una ruta o carpeta de nuestro ordenador, es decir, escoge una referencia. Allá donde haya establecido la ruta de trabajo (working directory) es donde guardará, leerá y buscará cualquier archivo.
Para saber donde se ha establecido la ruta de trabajo podemos preguntarselo a R con la función getwd() .
getwd()
[1] "/Users/adri/Desktop"
En mi caso tengo la referencia en mi carpeta de Escritorio ("/Users/adri/Desktop"). Aquí es donde ha establecido campamento R, su puerta de entrada, por lo que cualquier archivo y/o carpeta que se encuentre por debajo de esta ruta podrá ser encontrado por R. Pero que pasaría si quisiese encontrar un archivo que por ejemplo está en mi carpeta “adri” justo por encima de “Desktop”. Si este fuese el caso, R no podría encontrarlo dado que estaría fuera de la casa que ha elegido R para trabajar. Para que R no tuviese ningun problema a encontrar el archivo que se situa en la carpeta “adri” podríamos cambiar la ruta de trabajo directamente a la carpeta con el comando setwd()
setwd('/Users/adri/')
getwd()
[1] "/Users/adri"
También podremos hacerlo de manera “manual”. Basta navegar hasta la venta de Sesión (“Session”) y en la opción de “Set Working Directory” podemos elegir la ruta de trabajo.
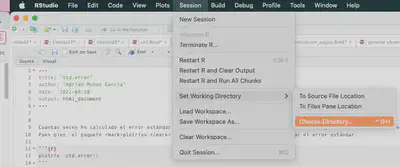
¿Todo esto quiere decir que siempre tengo que tener una ruta de trabajo que englobe el archivo que quiero importar? No. Ahora cuando revisemos las opciones de importación veremos que podemos definir directamente la ruta donde se encuentre el archivo. Sin embargo , entender la lógica de las sesiones y rutas de trabajo son fundamentales para entender como funciona R y resolver problemas que puedan surgir.
Importación de Datos
Importacion de objetos guardados previamente en R
Antes de pasar a importar datos con extensiones como .txt, .csv o .xlsx (Excel), creo que lo más sensato es aprender a importar datos que han sido previamente guardados en R.
¿Os acordaís que podemos almacenar objetos en nuestro Enviroment? Pues bien, todo ese espacio de trabajo podemos guardarlo y cargarlo. El espacio de trabajo tendrá extension .RData. Existen dos maneras tanto de guardar como de cargar los datos: Por código o através de atajos.
Guardar datos
- Codigo
save.image(file = "nombre.RData") # Guardamos como el nombre que querramos, sin especificar ruta # Si no especificamos la ruta, el archivo se guardará en nuestra ruta de trabajo getwd() save.image(file = "~/Desktop/nombre.RData") # Especifíco ruta- Atajo
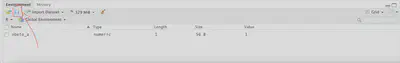
En la misma pestaña de Environment podemos clickar en el icono del disquete y elegir la ruta donde queremos guardar el archivo
Importar datos
- Codigo
load(file = "nombre.RData") load(file = "~/Desktop/nombre.RData") # Especifíco ruta si el archivo no se encuentra dentro de mi ruta de trabajo definida.- Atajo
En la misma pestaña de Environment podemos clickar en el icono de la carpeta y elegir el archivo con extensión .RData que queremos importar

Importacion desde Excel
Las importaciones de bases de datos más comunes provienen de Excel, o bien con formato .xlsx, o con formato .csv. Lo más común es enfrentarnos a una importación de este estilo cuando empezamos cualquier proyecto.
XLSX
El formato nativo de excel se llama .xlsx (antiguamente era .xls). Para importar archivos de este tipo existe una libreria muy útil llamada readxl y su función read_xlsx() aunque el proprio R base también nos permite importar archivos de este tipo.
La librería requiere dos argumentos primordiales: El nombre del archivo y su ruta (si no se encuentra en nuestra ruta de trabajo) y el núnero de la hoja de Excel que queremos importar. Por si no estaís familiarizados, un archivo de Excel puede contener varias hojas de trabajo, por esta razón la función necesita saber que hoja importar. Este argumento se puede resolver pasando el nombre de la ficha o su índice, es decir, si solo hubiese una hoja podríamos igualar el parámetro sheet a 1.
readxl::read_xlsx(path = "" ,sheet = )
readxl::read_xlsx(path = "~/Documentos_clases/Medicina/grupo_A.xlsx",
sheet = 1) # Si no le doy un nombre no lo guardo!
grupo_a <- readxl::read_xlsx(path ="/Users/adri/Desktop/URJC/DOCENCIA/2021:2022/MEDICINA/SESION_1/Listas/grupo_A.xlsx",
sheet = 1)
CSV
Tal vez sea el formato más frecuente en las importaciones de datos. Csv es la abreviación de (comma-separated values) y es precisamente porque los datos se guardan en un formato plano separados por unos delimitadores (generalmente comas , o puntos y coma ;). La razón del extendido uso de este formato se debe a la comprensión (redución del tamaño) que supone guardar los datos en un formato plano.
Para importar este tipo de archivos es necesario conocer cual es el caracter que delimita los diferentes datos. Además, debemos prestar atención a la unidad decimal de los números. Frecuentemente las bases de datos provienen de Estados Unidos donde el simbolo decimal es el punto., mientras que en España solemos utilizar la coma , como caracter decimal. ¿Veis un potencial riesgo? Si la delimitación es una coma y el símbolo decimal puede ser también la coma, esto puede acarrear algún problema? Debemos acostumbrarnos que del mismo modo que utilizamos el inglés para los textos científicos deberíamos utilizar la notación científica con el mismo formato.
Resumiendo, para importar archivos con formato csv podemos utilizar la función de R base read.csv(). En la misma función debemos específicar cuatro parametros importantes:
- file: El nombre (y ruta, si procede) del archivo
- sep: El caracter que se marcado para separar los datos (e.g., “;”)
- header: Marcar con
TRUEsi la primer línea de datos contiene los encabezados (nombres de las columnas o variables) - dec: Caracter que marca el símbolo decimal (e.g., “.”)
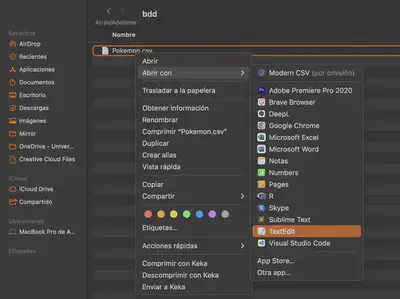
Pero claro… cómo podemos saber por ejemplo cuales son los caracteres que marcan la separación? Si no sabemos de antemano cuales son los caraceres podemos abrir el archivo csv en el bloc de notas (si estamos en Windows) o en TextEdit (para Mac). En este tipo de visores de texto simple podemos observar el verdadero formato csv y podremos ver los caracteres que han sido ajustados en ese archivo en específico. Para seleccionar un programa en concreto con el que abrir los archivos podemos hacer click izquierdo en el archivo y en la opción “Abrir con” elegir el programa que querramos.
read.csv(file = ,sep = ,header = ,dec = )
read.csv(file = "Desktop/archivo.csv",sep = ";",header = TRUE,dec = "." )
Limpia de nombres
Voy a introducir ya una función muy util para después de la importación de datos. Objetivamente no es necesario incluir el siguiente paso pero mi objetivo es acercaros las mejores prácticas desde el principio, o al menos, como me gusta trabajar a mi.
En muchas ocasiones los nombres de las variables vienen con algun espacio o juntando mayusculas y minusculas. De por sí no hay ningún problema, sin embargo, en la comodidad de escribir y trabajar con los datos, lidiar con nombres muy largos o con espacios puede ser muy pesado.
Existe un paquete con funciones muy interesantes para mejorar el flujo de trabajo Janitor . En este paquete se incluye una función que reduce y “limpia” los nombres de las variables para optimizar el trabajo. Adivinad el nombre abstracto con el que han bautizado a la función: clean_names()
Siempre que importo un df, me aseguro de correr en un segundo esta función. Fijaos en la diferencia. Con solo una función he simplificado los nombres. Ya no hay espacios, todo es en mínuscula y además la función ha sido inteligente como para sustituir la almohadilla # por “number”.
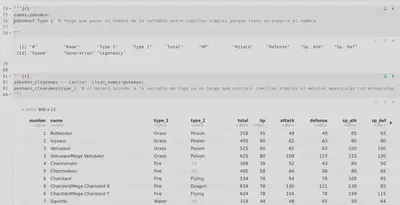
Esta práctica parece una tonteria pero mejora mucho la calidad de vida dentro de R. Con nombres accesibles, llamar a las variables resulta más cómodo. Con los nombres sin limpiar, para operar con la variable de tipo, deberíamos referirnos así: pokemon$Type 1y sin embargo, con un nombre sencilllo, en minúsculas y sin espacios: pokemon$type_1.
¡Acordaos de reescribir el df si haceís alguna modificación, sino, no se guarda!
pokemon_cleanames <- janitor::clean_names(pokemon)
pokemon_cleanames$type_1 # si quiero acceder a la variable de tipo ya no tengo que escribir comillas simples ni mezclar mayusculas con minusculas.
EJERCICIO importación csv
Os invito a probar a importar un archivo csv. Os dejo un enlace a una base de datos con extensión .csv de Pokémon. La base de datos la podeís descargar desde el link de la plataforma donde la encontré o directamente desde aquí
Aseguraros de hacer clik izquierdo en la pagina, luego en guardar como, y en añadir al nombre la extensión .csv.
VIDEO CON SOLUCIÓN
Exploración rápida datos
Por fin podemos trabajar o ver algo de datos. Lo primero, repito, lo primero que siempre debemos hacer al importar unos datos es echar un vistazo rápido a su estructura. Hay que ser igual de cotillas que Concha de “Aquí no hay quien viva”. Explorar en segundos los datos puede ahorrarnos problemas en los análisis. En primer lugar podemos detecar errores en la importación o errores intrinsecos de la base de datos. Por otro lugar, podemos empaparnos y comprender los datos. Los datos tienen un contexto y en los análisis e interpretaciones nunca podemos olvidarnos del contexto. Imaginad que estaís trabajando con una basde de datos de pacientes de un un hospital. En esa base de datos hay una variable llamada peso, y en el primer vistazo veís que hay un paciente con peso -5 ¿Ese dato está bien? Evidentmente ha habido un error, y lo sabemos porque el peso nunca puede ser negativo. Parece una estupidez, pero muchas veces cuando tratamos con datos nos olvidamos que los mismos se refieren a cosas reales, con lógica, y suprimimos el sentido común. En fin, continúo.
Hay varios modos de explorar rápidamente los datos. La opción más intuitiva es abrir lo mismos datos. De este modo puedo echar un vistazo muy rápido a los datos “crudos”, ver cuantas columnas tengo, como están organizados, si hay valores perdidos … en resumen, vemos superficialmente los datos, aunque no debemos subestimar este punto de vista

skimr::skim()
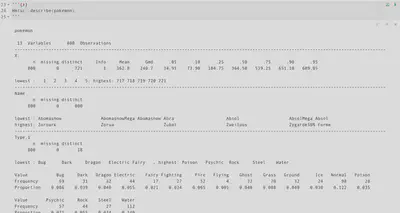
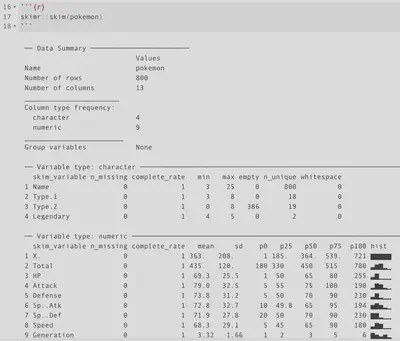
Mi método preferido para explorar rápidamente los datos es la función estrella del paquete skim skimr::skim()
De golpe podemos obtener un buen resumen de un dataframe En el resumen de los datos podemos observar el número de filas/observaciones y el número de columnas/variables. También podemos obtener unos descriptivos rápidos de las variables clasificadas en función de su tipo de variable (factor/character/numeric).
Podríamos ver hasta si una variable tiene valores perdidos, el rango de las valores en variables númericas, la cantidad de etiquetas diferentes en variables de texto, o si hay valores que no cuadran con la lógica de la variable.
Hay una cosa que me llamó la atención comparando las dos salidas de datos. Cuando llamé por primera vez al dataframe pensaba que había valores perdidos en la variable Type.2 al ver valores vacíos. Entonces … ¿No debería haber visto en la salida de
skim()que hubiese valores perdidos en la variable? Al ser una variable de texto, seguramente los valores en blanco precisamente estén en blanco, es decir, guardados como “”, por lo que existen en teoría aunque esten vacíos.
psych::describe()
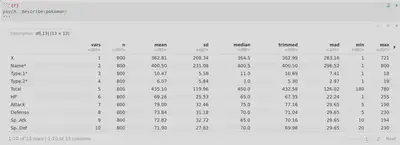
Otra solución útil es la función psych::describe() .
Esta función esta orientada a sacar los descriptivos de un conjunto de variables, sin embargo, también puede servir para explorar rápidamente los datos.
Sin embargo, debemos tener en cuenta que en un nuestro df podemos tener variables de texto por lo que no se podrían calcualar descriptivos per sè, y sin embargo, ahí están, marcadas con un *.
Hmisc::describe()
Una alternativa muy buena es utilizar la misma función describe() pero del paquete Hmisc .
Esta función es un poco más completa y ajusta los descriptivos en función del tipo de variable. Además, nos ofrece las frecuencias relativas o rangos para variables cuantitativas númericas, y un mapa resumido de valores distintos en variables de texto.